| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | ||||||
| 2 | 3 | 4 | 5 | 6 | 7 | 8 |
| 9 | 10 | 11 | 12 | 13 | 14 | 15 |
| 16 | 17 | 18 | 19 | 20 | 21 | 22 |
| 23 | 24 | 25 | 26 | 27 | 28 | 29 |
| 30 | 31 |
- Home Assistant
- 힐스테이트 광교산
- 국내주식
- 나스닥
- 취미생활
- 오블완
- 파이썬
- esp32
- 라즈베리파이
- 공모주
- RS-485
- Python
- 현대통신
- 매터
- ConnectedHomeIP
- MQTT
- Apple
- 월패드
- 애플
- Espressif
- 티스토리챌린지
- 배당
- 마이크로소프트
- matter
- 해외주식
- 홈네트워크
- homebridge
- raspberry pi
- Bestin
- 미국주식
- Today
- Total
YOGYUI
R pROC 패키지::ROC 커브 그리기, AUC 메트릭 계산하기 본문

빅데이터 분석기사 실기시험 R 개발환경에 ROCR 패키지가 제공되지 않아 대안을 찾아야했다
다행히 pROC 패키지가 제공되어서 간단한 사용법을 공부해봤다
(사실 AUC 구하는 수식 자체가 어려운게 아니라서 직접 구현해도 되긴 하지만... 패키지가 있다면 쓰는게 인지상정)
이진분류, 다중분류 모두 지원하니 케이스별로 테스트코드를 작성해보자 (함수명이 다르다)
유명한 공개 데이터셋 두 종류를 불러와 Train-Validation-Test 로 홀드아웃한 뒤, 랜덤포레스트 분류 모델 학습 후 검증 데이터, 테스트 데이터의 분류 결과를 살펴보자 (홀드아웃 비율은 8:1:1)
1. Binary Class Classfication
위스콘신 유방암 데이터로 이진분류 (메타데이터는 링크 참조)
분류대상 속성: BreastCancer$Class >> benign(양성) vs. malignant(악성)
library(mlbench)
data(BreastCancer)
# Replace attribute "Bare.nuclei" NA values to median
BreastCancer$Bare.nuclei[is.na(BreastCancer$Bare.nuclei)] <- median(as.numeric(BreastCancer$Bare.nuclei), na.rm = TRUE)
summary(BreastCancer$Bare.nuclei)
# Hold out
set.seed(210615)
library(caret)
idx1 <- caret::createDataPartition(BreastCancer$Class, p = 0.8)
df_train <- BreastCancer[idx1$Resample1, ]
df_temp <- BreastCancer[-idx1$Resample1,]
idx2 <- caret::createDataPartition(df_temp$Class, p = 0.5)
df_validation <- df_temp[idx2$Resample1, ]
df_test <- df_temp[-idx2$Resample1, ]
# Train Random Forest Classifier
library(randomForest)
model_rf <- randomForest(Class ~ .-Id, data = df_train)
library(pROC)
# Validation Dataset
pred_validation <- predict(model_rf, newdata = df_validation, type='response')
pred_validation_num <- as.numeric(pred_validation)
result_validation <- pROC::roc(df_validation$Class, pred_validation_num)predict 인자 type은 'response'로 해서 factor 자료형의 예측 결과 벡터(pred_validation)를 가져온 뒤 이를 numeric 자료형으로 변환(pred_validation_num)
pROC 패키지 roc 함수의 첫번째 인자로 정답라벨, 두번째 인자로 수치형 예측 결과 대입
ROC 커브는 plot.roc 함수를 사용하면 된다
pROC::plot.roc(result_test)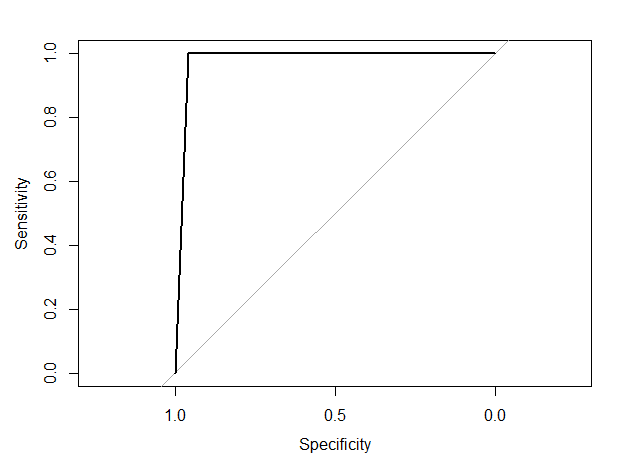
허접하게나마 ROC 커브를 그려볼 수 있다
가로축이 Specificity라서 반전이 되었다 (일반적으로 ROC Curve 가로축에는 False Positive Rate를 쓴다)
이는 legacy.axes = TRUE 인자를 집어넣어서 해결할 수 있다
pROC::plot.roc(result_validation, legacy.axes = TRUE)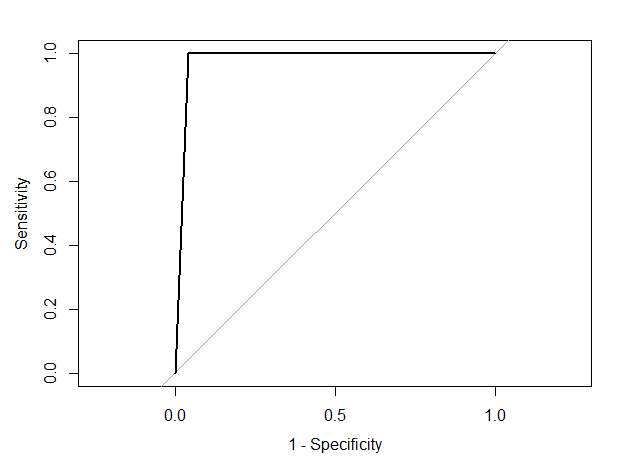
AUC값 가져오기는 간단하게 한줄로 해결할 수 있다
result_validation$auc
> Area under the curve: 0.9783
테스트 데이터의 Accuracy를 측정해보자
pred_test <- predict(model_rf, newdata = df_test, type='response')
same <- pred_test == df_test$Class
accuracy <- sum(same) / length(same)
accuracy
> [1] 0.9565217
2. Multi Class Classification
Glass Identification 데이터로 다중분류 (메타데이터는 링크 참조)
분류대상 속성: Glass$Type >> 1 ~ 7
rm(list=ls())
library(mlbench)
data(Glass)
# Hold out
set.seed(210615)
library(caret)
idx1 <- caret::createDataPartition(Glass$Type, p = 0.8)
df_train <- Glass[idx1$Resample1, ]
df_temp <- Glass[-idx1$Resample1, ]
idx2 <- caret::createDataPartition(df_temp$Type, p = 0.5)
df_validation <- df_temp[idx2$Resample1, ]
df_test <- df_temp[-idx2$Resample1, ]
# Train Random Forest Classifier
library(randomForest)
model_rf <- randomForest(Type ~ ., data = df_train)
library(pROC)
# Validation Dataset
pred_validation <- predict(model_rf, newdata = df_validation, type='response')
pred_validation_num <- as.numeric(pred_validation)
result_validation <- pROC::multiclass.roc(df_validation$Type, pred_validation_num)분류 대상 클래스가 3개 이상이라면 multiclass.roc 함수를 사용하면 된다 (사용법은 roc 함수와 동일)
roc 함수와는 달리 결과물을 다루는 방식이 조금 바뀐다 (분류 대상 클래스 수에 따라 roc 결과물이 여러개 생긴다)
result_validation$rocs
>
[[1]]
Call:
roc.default(response = response, predictor = predictor, levels = X, percent = percent, direction = ..1, auc = FALSE, ci = FALSE)
Data: predictor in 7 controls (response 1) < 8 cases (response 2).
Area under the curve not computed.
[[2]]
Call:
roc.default(response = response, predictor = predictor, levels = X, percent = percent, direction = ..1, auc = FALSE, ci = FALSE)
Data: predictor in 7 controls (response 1) < 2 cases (response 3).
Area under the curve not computed.
[[3]]
Call:
roc.default(response = response, predictor = predictor, levels = X, percent = percent, direction = ..1, auc = FALSE, ci = FALSE)
Data: predictor in 7 controls (response 1) < 1 cases (response 5).
Area under the curve not computed.
... (후략) ...
pROC::plot.roc(result_validation$rocs[[1]], legacy.axes = TRUE)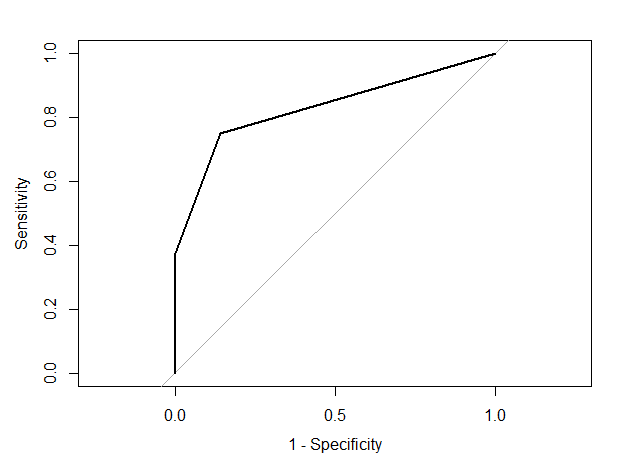
pROC::plot.roc(result_validation$rocs[[2]], legacy.axes = TRUE)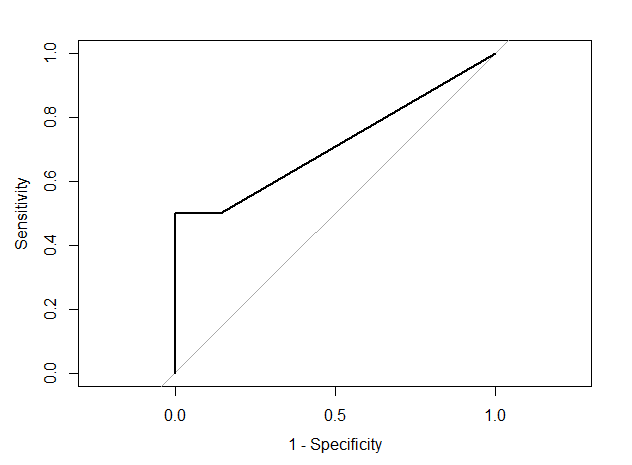
AUC는 이진 분류와 동일한 방식으로 얻을 수 있다
result_validation$auc
> Multi-class area under the curve: 0.8232
테스트 데이터의 Accuracy를 측정해보자
pred_test <- predict(model_rf, newdata = df_test, type='response')
same <- df_test$Type == pred_test
accuracy <- sum(same) / length(same)
accuracy
> [1] 0.7777778
'Software > R' 카테고리의 다른 글
| R caret::preProcess - 수치형 데이터 정규화, 표준화 (0) | 2021.06.17 |
|---|---|
| R 회귀분석 모델 성능판단 - RMSE, MAE, R squared (0) | 2021.06.16 |
| R NA 처리하기 (with dplyr) (0) | 2021.06.13 |
| R 스크립트 파일 경로 가져오기 (0) | 2021.06.05 |
| R 작업 경로(working directory, wd) Set/Get (0) | 2021.06.05 |




